| Enolase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
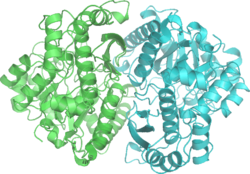 Dimer enolase yis[1] | |||||||||
| Pengenal pasti | |||||||||
| Nombor EC | 4.2.1.11 | ||||||||
| Nombor CAS | 9014-08-8 | ||||||||
| Pangkalan data | |||||||||
| IntEnz | Lihat IntEnz | ||||||||
| BRENDA | Entri BRENDA | ||||||||
| ExPASy | Lihat NiceZyme | ||||||||
| KEGG | Entri KEGG | ||||||||
| MetaCyc | Laluan metabolik | ||||||||
| PRIAM | Profil | ||||||||
| Struktur PDB | RCSB PDB PDBj PDBe PDBsum | ||||||||
| Ontologi gen | AmiGO / EGO | ||||||||
| |||||||||
| Enolase, domain terminal N | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Struktur dan mekanisme sinar-X enolase udang galah | |||||||||
| Pengenal pasti | |||||||||
| Simbol | Enolase_N | ||||||||
| Pfam | PF03952 | ||||||||
| Klan Pfam | CL0227 | ||||||||
| InterPro | IPR020811 | ||||||||
| PROSITE | PDOC00148 | ||||||||
| SCOP | 1els | ||||||||
| SUPERFAMILY | 1els | ||||||||
| |||||||||
| Enolase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| Pengenal pasti | |||||||||
| Simbol | Enolase | ||||||||
| Pfam | PF00113 | ||||||||
| InterPro | IPR000941 | ||||||||
| PROSITE | PDOC00148 | ||||||||
| |||||||||
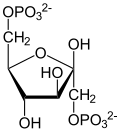
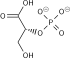
Fosfopiruvat hidratase, biasanya dikenali sebagai enolase, ialah metaloenzim (EC 4.2.1.11) yang memangkinkan penukaran 2-fosfogliserat (2-PG) kepada fosfoenolpiruvat (PEP), langkah kesembilan dan kedua terakhir bagi glikolisis. Tindak balas kimia ialah:
- 2-fosfo-D-gliserat fosfoenolpiruvat + H2O
Phosphopyruvate hydratase tergolong dalam keluarga liase, khususnya hidroliase yang membelah ikatan karbon-oksigen. Nama sistematik enzim ini ialah 2-fosfo-D-gliserat hidroliase (pembentuk fosfoenolpiruvat).
Tindak balas boleh diterbalikkan, dan bergantung kepada kepekatan persekitaran substrat.[3] pH optimum enzim manusia ialah 6.5.[4] Enolase terdapat dalam semua tisu dan organisma yang mampu melaksanakan glikolisis atau penapaian. Enzim ini ditemui oleh Lohmann dan Meyerhof pada tahun 1934,[5] dan sejak itu telah diasingkan daripada pelbagai sumber termasuk otot manusia dan eritrosit.[4] Pada manusia, kekurangan ENO1 dikaitkan dengan anemia hemolisis keturunan, manakala kekurangan ENO3 dikaitkan dengan penyakit penyimpanan glikogen jenis XIII.
Isozim
Pada manusia, terdapat tiga subunit enolase, α, β dan γ, masing-masing dikodkan oleh gen berasingan yang boleh bergabung untuk membentuk lima isoenzim yang berbeza: αα, αβ, αγ, ββ, dan γγ.[3][6] Tiga daripada isoenzim ini (semua homodimer) lebih kerap ditemui dalam sel manusia dewasa berbanding yang lain:
- αα atau enolase bukan neuron (NNE). Juga dikenali sebagai enolase 1. Ditemui dalam pelbagai tisu, termasuk hati, otak, buah pinggang, limpa dan adiposa. Ia hadir pada tahap tertentu dalam semua sel manusia normal.
- ββ atau enolase khusus otot (MSE). Juga dikenali sebagai enolase 3. Enzim ini sebahagian besarnya terhad kepada otot, di mana ia terdapat dalam tahap yang sangat tinggi dalam.
- γγ atau enolase khusus neuron (NSE). Juga dikenali sebagai enolase 2. Dinyatakan pada tahap yang sangat tinggi dalam neuron dan tisu saraf, di mana ia boleh menyumbang sebanyak 3% daripada jumlah protein larut. Ia dinyatakan pada tahap yang jauh lebih rendah dalam kebanyakan sel mamalia.
Struktur
Enolase ialah ahli superkeluarga enolase yang besar. Ia mempunyai berat molekul 82,000–100,000 dalton, bergantung kepada isoform.[3][4] Dalam alfa enolase manusia, dua subunit adalah antiselari dalam orientasi, dengan Glu20 daripada satu subunit membentuk ikatan ionik dengan Arg414 daripada subunit yang lain.[3] Setiap subunit mempunyai dua domain yang berbeza. Domain terminal N yang lebih kecil terdiri daripada tiga heliks α dan empat helaian β.[3][6] Domain terminal C yang lebih besar bermula dengan dua helaian β, diikuti dengan dua heliks α, dan berakhir dengan balang yang terdiri daripada helaian β berselang-seli dan heliks α yang disusun supaya helaian β dikelilingi oleh heliks α.[3][6] Struktur globul padat enzim terhasil daripada interaksi hidrofobik yang ketara antara kedua-dua domain ini.
Enolase ialah enzim yang sangat terpelihara, dengan lima sisa tapak aktif yang sangat penting untuk aktiviti. Jika dibandingkan dengan enolase jenis liar, enolase mutan yang berbeza sama ada pada residu Glu168, Glu211, Lys345 atau Lys396 mempunyai tahap aktiviti yang dihadkan dengan faktor 105.[3] Satu lagi hal, perubahan yang mempengaruhi His159 meninggalkan mutan dengan hanya 0.01% daripada aktiviti pemangkinnya.[3] Bahagian penting enolase ialah dua kofaktor Mg2+ dalam tapak aktif yang berfungsi untuk menstabilkan cas negatif dalam substrat.[3][6]
Baru-baru ini, fungsi sampingan beberapa enolase seperti interaksi dengan plasminogen, telah menarik minat terhadap gelung pemangkinan enzim dan kepelbagaian strukturnya.[7][8]
Mekanisme

Menggunakan prob isotop, mekanisme keseluruhan untuk menukar 2-PG kepada PEP dicadangkan sebagai tindak balas penyingkiran E1cB yang melibatkan perantaraan karbanion.[9] Mekanisme terperinci berikut adalah berdasarkan kajian struktur kristal dan kinetik.[3][10][11][12][13][14][15] Apabila substrat, 2-fosfogliserat, mengikat α-enolase, kumpulan karboksilnya menyelaras dengan dua kofaktor ion magnesium dalam tapak aktif. Ini menstabilkan cas negatif pada oksigen ternyahproton sambil meningkatkan keasidan hidrogen alfa. Lys345 Enolase menyahprotonasi hidrogen alfa, dan cas negatif yang terhasil distabilkan oleh resonans kepada oksigen karboksilat dan oleh kofaktor ion magnesium. Berikutan penciptaan perantaraan karbanion, hidroksida pada C3 disingkirkan sebagai air dengan bantuan Glu211, dan PEP terbentuk.
Selain itu, perubahan konformasi berlaku dalam enzim yang membantu pemangkinan. Dalam α-enolase manusia, substrat diputar ke dalam kedudukan apabila mengikat enzim disebabkan oleh interaksi dengan dua ion magnesium pemangkin, Gln167 dan Lys396. Pergerakan gelung Ser36 ke His43, Ser158 ke Gly162, dan Asp255 ke Asn256 membolehkan Ser39 menyelaras dengan Mg2+ lalu menutup tapak aktif. Sebagai tambahan kepada penyelarasan dengan ion magnesium pemangkin, pKa hidrogen alfa substrat juga diturunkan disebabkan oleh protonasi kumpulan fosforil oleh His159 dan kedekatannya dengan Arg374. Arg374 juga menyebabkan Lys345 dalam tapak aktif ternyahproton, yang menetapkan Lys345 dalam peranannya dalam mekanisme.
Kegunaan diagnosis
Dalam eksperimen perubatan baru-baru ini, kepekatan enolase telah diambil sampel dalam percubaan untuk mendiagnosis keadaan tertentu dan keterukan mereka. Contohnya, kepekatan enolase yang lebih tinggi dalam cecair serebrospinal lebih kuat berkorelasi dengan astrositoma gred rendah berbanding enzim lain yang diuji (aldolase, piruvat kinase, kreatina kinase dan laktat dehidrogenase).[16] Kajian yang sama menunjukkan bahawa kadar pertumbuhan tumor terpantas berlaku pada pesakit dengan tahap enolase CSF tertinggi. Peningkatan tahap enolase juga telah dikenal pasti pada pesakit yang telah mengalami infarksi miokardium atau kemalangan serebrovaskular baru-baru ini. Ada kesimpulan bahawa tahap enolase khusus neuron CSF, serum NSE, dan kreatina kinase (jenis BB) boleh menjadi petunjuk dalam penilaian prognosis mangsa serangan jantung.[17] Kajian lain telah memberi tumpuan kepada nilai prognostik nilai NSE dalam mangsa kemalangan serebrovaskular.[18]
Autoantibodi kepada alfa-enolase dikaitkan dengan artritis reumatoid[19] dan sindrom jarang jumpa, ensefalopati Hashimoto.[20]
Perencat
Perencat molekul kecil enolase telah disintesis sebagai probe kimia (substrat-analog) mekanisme pemangkin enzim dan baru-baru ini, telah disiasat sebagai rawatan berpotensi dalam kanser dan penyakit berjangkit.[21][22] Kebanyakan perencat mempunyai sifat pengkelat logam dan mengikat enzim melalui interaksi dengan atom magnesium struktur, Mg(A).[23][24] Sebatian yang paling mujarab ialah fosfonoasetohidroksamat[24] yang memiliki mempunyai pertalian pM untuk enzim dalam bentuk tidak terprotonasi. Ia mempunyai persamaan struktur dengan perantara pemangkin yang dianggap, antara PEP dan 2-PG. Percubaan telah dibuat untuk menggunakan perencat ini sebagai ubat antitripanosom,[25] dan lebih baru-baru ini, sebagai agen antikanser, khususnya, dalam glioblastoma kekurangan enolase disebabkan oleh penghapusan homozigot gen ENO1 sebagai sebahagian daripada lokus penindas tumor 1p36 (kematian sintetik).[26] Antibiotik fosfonat produk semula jadi, SF2312 (CAS 107729-45-3) yang aktif terhadap bakteria positif dan negatif Gram, terutamanya dalam keadaan anaerob,[27] ialah perencat potensi tinggi enolase 4zcw yang mengikat dengan cara yang serupa dengan fosfonoasetohidroksamat, 4za0.[28] SF2312 menghalang aktiviti enolase dalam kedua-dua asal eukariot dan prokariot,[29] mencerminkan pemuliharaan evolusi enolase yang kuat dan asal purba laluan glikolisis. SF2312 ialah molekul kiral dengan hanya 3S-enantiomer yang menunjukkan aktiviti perencatan enolase dan aktiviti biologi terhadap bakteria.[30] Baru-baru ini, terbitan SF2312 yang dipanggil HEX, dan prodrugnya, POMHEX, ditunjukkan menggunakan aktiviti antineoplastik terhadap glioma yang dipadamkan ENO1 dalam model tetikus ortotopik intrakranium praklinikal.[31] Pengikat alosterik, ENOblock,[22] pada mulanya digambarkan sebagai perencat enolase, tetapi kemudiannya ditunjukkan tidak sebenarnya menghalang enzim, sebaliknya, mengganggu ujian enzim enolase in vitro.[32] ENOblock didapati mengubah penyetempatan sel enolase, mempengaruhi fungsi sekunder bukan glikolisis seperti peraturan transkripsi.[33] Analisis seterusnya menggunakan ujian komersial juga menunjukkan bahawa ENOblock boleh menghalang aktiviti enolase dalam konteks biologi, seperti sel dan tisu haiwan.[33] Metilglioksal juga telah digambarkan sebagai perencat enolase manusia.[34]
Perencat enolase analog keadaan peralihan tapak aktif telah diterokai secara praklinikal untuk rawatan pelbagai patogen mikrob, serta dalam onkologi ketepatan untuk tumor dengan penghapusan homozigot 1p36 yang kekurangan ENO1.[31][35][36][37][38][39][40]
Rujukan
- ^ PDB: 2ONE; Zhang E, Brewer JM, Minor W, Carreira LA, Lebioda L (October 1997). "Mechanism of enolase: the crystal structure of asymmetric dimer enolase-2-phospho-D-glycerate/enolase-phosphoenolpyruvate at 2.0 A resolution". Biochemistry. 36 (41): 12526–12534. doi:10.1021/bi9712450. PMID 9376357.
- ^ PDB: 2XSX; Vollmar M, Krysztofinska E, Chaikuad A, Krojer T, Cocking R, Vondelft F, Bountra C, Arrowsmith CH, Weigelt J, Edwards A, Yue WW, Oppermann U (2010). "Crystal structure of human beta enolase ENOB". Protein Data Bank.
- ^ a b c d e f g h i j "Multifunctional alpha-enolase: its role in diseases". Cellular and Molecular Life Sciences. 58 (7): 902–920. June 2001. doi:10.1007/pl00000910. PMID 11497239.
- ^ a b c "Purification and properties of enolase of human erythrocytes". International Journal of Biochemistry. 5 (11–12): 845–852. November 1974. doi:10.1016/0020-711X(74)90119-0.
|hdl-access=requires|hdl=(bantuan) - ^ Lohman, K; Meyerhof, O (1934). "Über die enzymatische umwandlung von phosphoglyzerinsäure in brenztraubensäure und phosphorsäure" [Enzymatic transformation of phosphoglyceric acid into pyruvic and phosphoric acid]. Biochemische Zeitschrift (dalam bahasa Jerman). 273: 60–72.
- ^ a b c d "Molecular structure of the human muscle-specific enolase gene (ENO3)". The Biochemical Journal. 275 ( Pt 2) (Pt 2): 427–433. April 1991. doi:10.1042/bj2750427. PMC 1150071. PMID 1840492.
- ^ "Plasmin(ogen)-binding alpha-enolase from Streptococcus pneumoniae: crystal structure and evaluation of plasmin(ogen)-binding sites". Journal of Molecular Biology. 343 (4): 997–1005. October 2004. doi:10.1016/j.jmb.2004.08.088. PMID 15476816.
- ^ "Crystal structure of an efficacious gonococcal adherence inhibitor: an enolase from Lactobacillus gasseri". FEBS Letters. 588 (14): 2212–2216. June 2014. doi:10.1016/j.febslet.2014.05.020. PMID 24859038.
- ^ "Isotopic probes of the enolase reaction mechanism". J Biol Chem. 240 (14): 4586–93. 1971. doi:10.1016/S0021-9258(18)62051-4.
- ^ "Toward identification of acid/base catalysts in the active site of enolase: comparison of the properties of K345A, E168Q, and E211Q variants". Biochemistry. 35 (5): 1692–1699. February 1996. doi:10.1021/bi952186y. PMID 8634301.
- ^ "Structural and mechanistic studies of enolase". Current Opinion in Structural Biology. 6 (6): 736–743. December 1996. doi:10.1016/S0959-440X(96)80002-9. PMID 8994873.
- ^ "Octahedral coordination at the high-affinity metal site in enolase: crystallographic analysis of the MgII--enzyme complex from yeast at 1.9 A resolution". Biochemistry. 34 (13): 4325–4330. April 1995. doi:10.1021/bi00013a022. PMID 7703246.
- ^ "Chelation of serine 39 to Mg2+ latches a gate at the active site of enolase: structure of the bis(Mg2+) complex of yeast enolase and the intermediate analog phosphonoacetohydroxamate at 2.1-A resolution". Biochemistry. 33 (31): 9333–9342. August 1994. doi:10.1021/bi00197a038. PMID 8049235.
- ^ "A carboxylate oxygen of the substrate bridges the magnesium ions at the active site of enolase: structure of the yeast enzyme complexed with the equilibrium mixture of 2-phosphoglycerate and phosphoenolpyruvate at 1.8 A resolution". Biochemistry. 35 (14): 4349–4358. April 1996. doi:10.1021/bi952859c. PMID 8605183.
- ^ "X-ray structure and catalytic mechanism of lobster enolase". Biochemistry. 34 (39): 12513–12523. October 1995. doi:10.1021/bi00039a005. PMID 7547999.
- ^ "Levels of enolase and other enzymes in the cerebrospinal fluid as indices of pathological change". Journal of Neurology, Neurosurgery, and Psychiatry. 44 (12): 1129–1135. December 1981. doi:10.1136/jnnp.44.12.1129. PMC 491233. PMID 7334408.
- ^ "Neurological outcome after out-of-hospital cardiac arrest. Prediction by cerebrospinal fluid enzyme analysis". Archives of Neurology. 46 (7): 753–756. July 1989. doi:10.1001/archneur.1989.00520430047015. PMID 2742544.
- ^ "Cerebrospinal fluid enolase in stroke". Journal of Neurology, Neurosurgery, and Psychiatry. 47 (7): 724–729. July 1984. doi:10.1136/jnnp.47.7.724. PMC 1027902. PMID 6747647.
- ^ "Antibodies to citrullinated alpha-enolase peptide 1 are specific for rheumatoid arthritis and cross-react with bacterial enolase". Arthritis and Rheumatism. 58 (10): 3009–3019. October 2008. doi:10.1002/art.23936. PMID 18821669. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "Autoantibodies against the amino terminal of alpha-enolase are a useful diagnostic marker of Hashimoto's encephalopathy". Journal of Neuroimmunology. 162 (1–2): 130–136. May 2005. doi:10.1016/j.jneuroim.2005.02.004. PMID 15833368. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "Reaction intermediate analogues for enolase". Biochemistry. 23 (12): 2779–2786. June 1984. doi:10.1021/bi00307a038. PMID 6380574.
- ^ a b "A unique small molecule inhibitor of enolase clarifies its role in fundamental biological processes". ACS Chemical Biology. 8 (6): 1271–1282. 2 April 2013. doi:10.1021/cb300687k. PMID 23547795. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "Structure of the bis divalent cation complex with phosphonoacetohydroxamate at the active site of enolase". Biochemistry. 31 (31): 7166–7173. August 1992. doi:10.1021/bi00146a020. PMID 1322695.
- ^ a b "Catalytic metal ion binding in enolase: the crystal structure of an enolase-Mn2+-phosphonoacetohydroxamate complex at 2.4-A resolution". Biochemistry. 33 (20): 6295–6300. May 1994. doi:10.1021/bi00186a032. PMID 8193144.
- ^ "Passenger deletions generate therapeutic vulnerabilities in cancer". Nature. 488 (7411): 337–342. August 2012. Bibcode:2012Natur.488..337M. doi:10.1038/nature11331. PMC 3712624. PMID 22895339. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "Studies on a new phosphonic acid antibiotic, SF-2312". Sci Rep Meiji Seika Kaisha. 25: 12–17. 1986.
- ^ "SF2312 is a natural phosphonate inhibitor of enolase". Nature Chemical Biology. 12 (12): 1053–1058. December 2016. doi:10.1038/nchembio.2195. PMC 5110371. PMID 27723749. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "Functional and structural basis of E. coli enolase inhibition by SF2312: a mimic of the carbanion intermediate". Scientific Reports. 9 (1): 17106. November 2019. Bibcode:2019NatSR...917106K. doi:10.1038/s41598-019-53301-3. PMC 6863902. PMID 31745118. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "The 3S Enantiomer Drives Enolase Inhibitory Activity in SF2312 and Its Analogues". Molecules. 24 (13): 2510. July 2019. doi:10.3390/molecules24132510. PMC 6651268. PMID 31324042. Unknown parameter
|displayauthors=ignored (bantuan) - ^ a b "An enolase inhibitor for the targeted treatment of ENO1-deleted cancers". Nature Metabolism. 2 (12): 1413–1426. December 2020. doi:10.1038/s42255-020-00313-3. PMC 7744354. PMID 33230295. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "ENOblock Does Not Inhibit the Activity of the Glycolytic Enzyme Enolase". PLOS ONE. 11 (12): e0168739. 28 December 2016. Bibcode:2016PLoSO..1168739S. doi:10.1371/journal.pone.0168739. PMC 5193436. PMID 28030597.
- ^ a b "ENOblock, a unique small molecule inhibitor of the non-glycolytic functions of enolase, alleviates the symptoms of type 2 diabetes". Scientific Reports. 7: 44186. March 2017. Bibcode:2017NatSR...744186C. doi:10.1038/srep44186. PMC 5341156. PMID 28272459. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "Inhibition of human muscle-specific enolase by methylglyoxal and irreversible formation of advanced glycation end products". Journal of Enzyme Inhibition and Medicinal Chemistry. 24 (2): 356–364. April 2009. doi:10.1080/14756360802187679. PMID 18830874.
- ^ "Targeting Host Glycolysis as a Strategy for Antimalarial Development". Frontiers in Cellular and Infection Microbiology. 11: 730413. 16 September 2021. doi:10.3389/fcimb.2021.730413. PMC 8482815 Check
|pmc=value (bantuan). PMID 34604112 Check|pmid=value (bantuan). Unknown parameter|displayauthors=ignored (bantuan) - ^ "Structure-guided microbial targeting of antistaphylococcal prodrugs". eLife. 10: e66657. July 2021. doi:10.7554/eLife.66657. PMC 8318587 Check
|pmc=value (bantuan). PMID 34279224. Unknown parameter|displayauthors=ignored (bantuan) - ^ "Antimicrobial Prodrug Activation by the Staphylococcal Glyoxalase GloB". ACS Infectious Diseases. 6 (11): 3064–3075. November 2020. doi:10.1021/acsinfecdis.0c00582. PMC 8543975 Check
|pmc=value (bantuan). PMID 33118347. Unknown parameter|displayauthors=ignored (bantuan) - ^ "Enhancer Reprogramming Confers Dependence on Glycolysis and IGF Signaling in KMT2D Mutant Melanoma". Cell Reports. 33 (3): 108293. October 2020. doi:10.1016/j.celrep.2020.108293. PMC 7649750. PMID 33086062. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "Functional and structural basis of E. coli enolase inhibition by SF2312: a mimic of the carbanion intermediate". Scientific Reports. 9 (1): 17106. November 2019. Bibcode:2019NatSR...917106K. doi:10.1038/s41598-019-53301-3. PMC 6863902. PMID 31745118. Unknown parameter
|displayauthors=ignored (bantuan) - ^ "The 3S Enantiomer Drives Enolase Inhibitory Activity in SF2312 and Its Analogues". Molecules. 24 (13): 2510. July 2019. doi:10.3390/molecules24132510. PMC 6651268. PMID 31324042. Unknown parameter
|displayauthors=ignored (bantuan)